LIMSwiki

Protein adalah kelompok biomolekul berukuran besar yang terbentuk dari satu rantai panjang asam amino atau lebih. Protein memiliki banyak fungsi dalam makhluk hidup, di antaranya mempercepat reaksi-reaksi metabolisme, mereplikasi DNA, menanggapi rangsangan, memberi bentuk sel dan tubuh, dan memindahkan molekul dari satu lokasi ke lokasi lain. Perbedaan utama antara satu protein dan protein lainnya adalah urutan asam amino-asam aminonya, yang ditentukan oleh urutan nukleotida dari gen-gennya, dan biasanya menyebabkan lipatan protein menjadi struktur tiga dimensi khusus yang sesuai dengan fungsinya.
Sejumlah asam amino membentuk rantai lurus yang disebut polipeptida. Suatu protein terdiri dari minimum satu polipeptida panjang. Polipeptida pendek (dengan kurang dari 20–30 asam amino) biasanya tidak dianggap sebagai protein, tetapi disebut molekul peptida atau oligopeptida. Masing-masing asam amino dalam protein terikat ke asam amino di dekatnya oleh ikatan peptida. Urutan asam amino dalam protein ditentukan oleh urutan gen yang disandi dalam kode genetik. Secara umum, kode genetik menghasilkan 20 asam amino standar, meskipun beberapa organisme memiliki asam amino tambahan. Tak lama setelah atau bahkan selama sintesis, residu dalam protein sering dimodifikasi secara kimiawi melalui proses modifikasi pascatranslasi yang mengubah sifat fisik dan kimia, lipatan, stabilitas, aktivitas, dan fungsi protein. Beberapa protein memiliki gugus nonpeptida (bukan asam amino), yang dapat disebut kofaktor dan gugus prostetik. Beberapa protein juga dapat bekerja sama untuk menjalankan fungsi tertentu, dan kelompok seperti ini sering membentuk kompleks protein yang stabil.
Begitu terbentuk, protein hanya ada untuk jangka waktu tertentu lalu didegradasi dan didaur ulang dalam sel melalui proses pergantian protein. Umur protein diukur berdasarkan waktu paruhnya dan mencakup rentang yang panjang. Protein bisa berumur beberapa menit hingga beberapa tahun dengan umur rata-rata 1–2 hari dalam sel mamalia. Protein yang abnormal atau salah lipat akan terdegradasi lebih cepat, baik karena ditargetkan untuk dihancurkan atau karena tidak stabil.
Bersama dengan biomolekul raksasa lainnya seperti polisakarida dan asam nukleat, protein merupakan bagian esensial dari organisme dan terlibat dalam hampir seluruh proses di dalam sel. Sebagian protein adalah enzim yang berfungsi sebagai katalis dalam reaksi-reaksi biokimia dan bersifat vital untuk metabolisme. Sebagian protein memiliki fungsi pembentuk atau penguat, misalnya protein aktin dan miosin dalam otot dan protein-protein dalam sitoskeleton. Protein-protein lainnya memiliki peran penting dalam persinyalan sel, respons imun, adhesi sel, dan siklus sel. Hewan memerlukan protein dalam makanannya untuk memperoleh asam amino esensial yang tidak bisa disintesis di dalam tubuh. Sistem pencernaan memecah protein dari makanan untuk dapat digunakan dalam metabolisme.
Protein dapat dimurnikan dari komponen seluler lainnya menggunakan berbagai teknik seperti ultrasentrifugasi, presipitasi, elektroforesis, dan kromatografi. Rekayasa genetika memungkinkan sejumlah metode untuk memfasilitasi pemurnian ini. Metode yang biasa dipakai untuk mempelajari struktur dan fungsi protein yaitu imunohistokimia, mutagenesis terarah-lokasi, kristalografi sinar-X, resonansi magnetik inti, dan spektrometri massa.
Protein merupakan suatu zat makanan yang amat penting bagi tubuh, karena zat ini berfungsi sebagai bahan bakar, zat pembangun dan pengatur. Dan fungsi utama dari protein adalah membentuk jaringan baru dan mempertahankan jaringan yang telah ada. Mutu protein dalam bahan makanan yang di konsumsi manusia yang akan di serap oleh usus dalam bentuk asam amino [1]
Sejarah dan etimologi
Protein dikenali sebagai kelompok biomolekul pada abad kedelapan belas oleh Antoine Fourcroy dan lain-lain, yang dicirikan oleh kemampuannya untuk melakukan koagulasi atau flokulasi di bawah perlakuan dengan panas atau asam.[2] Contoh yang tercatat pada saat itu adalah albumin dari putih telur, albumin dalam serum darah, fibrin, dan gluten gandum.
Protein pertama kali dijelaskan oleh kimiawan Belanda Gerardus Johannes Mulder dan dinamai oleh ahli kimia Swedia Jöns Jacob Berzelius pada tahun 1838.[3][4] Mulder melakukan analisis unsur terhadap protein umum dan menemukan bahwa hampir semua protein memiliki rumus empiris yang sama, yaitu C400H620N100O120P1S1.[5] Ia sampai pada kesimpulan yang salah bahwa mereka mungkin terdiri dari satu jenis molekul (sangat besar). Istilah "protein" untuk menggambarkan molekul-molekul ini diajukan oleh rekan Mulder, Berzelius; protein berasal dari kata Yunani πρώτειος (proteios), yang berarti "primer",[6] "di depan", atau "berdiri di depan",[7] ditambah akhiran -in. Mulder selanjutnya mengidentifikasi produk degradasi protein seperti asam amino leusin yang ia temukan dengan berat molekul (hampir benar) 131 Da.[5] Sebelum "protein", nama lainnya telah digunakan, seperti "albumin" atau "bahan albumin" (Eiweisskörper, dalam bahasa Jerman).[8]
Ilmuwan nutrisi awal seperti Carl von Voit dari Jerman percaya bahwa protein adalah nutrisi terpenting untuk menjaga struktur tubuh karena secara umum diyakini bahwa "daging membuat daging."[9] Karl Heinrich Ritthausen memperluas bentuk protein yang diketahui dengan mengidentifikasi asam glutamat. Di Stasiun Percobaan Pertanian Connecticut, tinjauan terperinci tentang protein nabati dikumpulkan oleh Thomas Burr Osborne. Ia bekerja dengan Lafayette Mendel dan menerapkan hukum minimum Liebig dalam memberi makan tikus laboratorium, sehingga adanya asam amino esensial pun diketahui. Pekerjaan ini dilanjutkan dan dikomunikasikan oleh William Cumming Rose. Pemahaman tentang protein sebagai polipeptida muncul melalui karya Franz Hofmeister dan Hermann Emil Fischer pada tahun 1902.[10][11] Peran sentral protein sebagai enzim dalam organisme hidup tidak sepenuhnya diapresiasi sampai tahun 1926 ketika James B. Sumner menunjukkan bahwa enzim urease sebenarnya adalah protein.[12]
Kesulitan dalam memurnikan protein dalam jumlah besar membuat para ahli biokimia protein awal sangat sulit mempelajarinya. Oleh karena itu, penelitian awal difokuskan pada protein yang dapat dimurnikan dalam jumlah besar, misalnya dari darah, putih telur, berbagai racun, dan enzim pencernaan/metabolik yang diperoleh dari rumah pemotongan hewan. Pada 1950-an, Armor Hot Dog Co. memurnikan 1 kg ribonuklease A dari pankreas sapi murni dan menyediakannya secara gratis bagi para ilmuwan; gerakan ini membantu ribonuklease A menjadi target utama studi biokimia selama beberapa dekade berikutnya.[5]

Linus Pauling dianggap sukses dalam memperkirakan struktur sekunder protein biasa berdasarkan ikatan hidrogen, sebuah ide yang pertama kali dikemukakan oleh William Astbury pada tahun 1933.[13] Belakangan, karya Walter Kauzmann tentang denaturasi,[14][15] yang sebagian didasarkan pada penelitian sebelumnya oleh Kaj Linderstrøm-Lang,[16] memberi pemahaman tentang pelipatan protein dan struktur yang dimediasi oleh interaksi hidrofobik.
Protein pertama yang diurutkan adalah insulin, oleh Frederick Sanger, pada 1949. Sanger dengan tepat menentukan urutan asam amino insulin sehingga secara meyakinkan menunjukkan bahwa protein terdiri dari polimer linier asam amino alih-alih rantai bercabang, koloid, atau siklol.[17] Ia memenangkan Hadiah Nobel untuk pencapaian ini pada 1958.[18]
Struktur protein pertama yang diketahhui adalah hemoglobin dan mioglobin, masing-masing oleh Max Perutz dan Sir John Cowdery Kendrew, pada tahun 1958.[19][20] Hingga 2017, Bank Data Protein memiliki lebih dari 126.060 struktur protein dengan resolusi atomik.[21] Baru-baru ini, mikroskop krio-elektron terhadap kumpulan makromolekul besar[22] dan prediksi struktur protein komputasional terhadap domain protein kecil[23] adalah dua metode yang mendekati resolusi atomik.
Jumlah protein yang disandi dalam genom
Jumlah protein yang disandi dalam genom secara kasar sesuai dengan jumlah gen (walaupun mungkin ada sejumlah besar gen yang menyandi RNA protein, misalnya RNA ribosomal). Virus biasanya menyandikan beberapa ratus protein, arkea dan bakteri beberapa ratus hingga beberapa ribu, sementara eukariota biasanya menyandikan beberapa ribu hingga puluhan ribu protein (lihat ukuran genom untuk daftar contoh).
Biokimia


Protein merupakan biomolekul yang sangat besar atau makrobiopolimer yang tersusun dari monomer berupa asam amino. Ada 20 asam amino standar yang membentuk asam amino (disebut asam amino proteinogenik); masing-masing terdiri dari sebuah karbon alfa yang berikatan dengan sebuah gugus amino (–NH2), sebuah gugus karboksil (–COOH), sebuah atom hidrogen (H), dan rantai samping (disebut sebagai "R"). Gugus "R" inilah yang menjadikan setiap asam amino berbeda dan sifat rantai samping ini akan memengaruhi keseluruhan suatu protein. Hanya prolina yang berbeda dari struktur dasar ini karena mengandung cincin yang tidak biasa pada gugus amina ujung-N, yang memaksa gugus amida CO–NH menjadi konformasi tetap.[24] Rantai samping asam amino standar, yang dirinci dalam daftar asam amino standar, memiliki beragam struktur dan sifat kimiawi. Struktur tiga dimensi dan reaktivitas kimia suatu protein ditentukan oleh efek gabungan dari semua rantai samping asam amino dalam protein tersebut.[25] Semua asam amino dalam rantai polipeptida saling terhubung oleh ikatan peptida melalui sintesis dehidrasi. Setelah terhubung dalam rantai protein, asam amino individual disebut residu, sedangkan rangkaian atom karbon, nitrogen, dan oksigen yang terkait disebut rantai utama atau tulang punggung protein.[26]
Istilah protein, polipeptida, dan peptida agak ambigu dan dapat tumpang tindih artinya. Protein umumnya digunakan untuk merujuk pada molekul biologis lengkap dalam konformasi yang stabil, sedangkan peptida umumnya merujuk pada oligomer asam amino pendek yang sering kali tidak memiliki struktur tiga dimensi yang stabil. Namun, batas antara keduanya tidak ditentukan dengan baik dan biasanya berkisar antara 20–30 residu. Polipeptida dapat merujuk pada rantai linier tunggal asam amino, biasanya berapa pun panjangnya, tetapi sering kali menyiratkan tidak memiliki konformasi yang tetap.
Interaksi
Protein dapat berinteraksi dengan banyak jenis molekul, termasuk dengan protein lain, dengan lipid, dengan karbohidrat, dan dengan DNA.[27][28][29][30]
Kelimpahan dalam sel
Diperkirakan bahwa bakteri berukuran rata-rata mengandung sekitar dua juta protein per sel (misalnya Escherichia coli dan Staphylococcus aureus). Bakteri yang lebih kecil, seperti Mycoplasma atau spiroket mengandung lebih sedikit protein, sekitar 50.000 hingga 1 juta. Sel eukariota berukuran lebih besar sehingga mengandung lebih banyak protein. Misalnya, sel khamir Saccharomyces cerevisiae diperkirakan mengandung sekitar 50 juta protein dan sel manusia sekitar 1 hingga 3 miliar.[31] Konsentrasi salinan protein individual berkisar dari beberapa molekul per sel hingga 20 juta per sel.[32] Tidak semua gen yang menyandi protein diekspresikan di sebagian besar sel dan jumlahnya bergantung pada beberapa hal, seperti jenis sel dan rangsangan eksternal. Misalnya, dari sekitar 20.000 protein yang disandi oleh genom manusia, hanya 6.000 yang terdeteksi dalam sel limfoblastoid.[33]
Sintesis
Biosintesis


Protein dirakit dari sejumlah asam amino menggunakan informasi yang disandi dalam gen. Setiap protein memiliki urutan asam amino uniknya sendiri yang ditentukan oleh urutan nukleotida dari gen yang menyandi protein ini. Kode genetik adalah satu set berupa tiga nukleotida yang disebut kodon dan setiap kombinasi tiga nukleotida menunjukkan asam amino, misalnya AUG (adenina–urasil–guanina) adalah kode untuk metionin. Karena DNA mengandung empat nukleotida, jumlah total kodon yang mungkin adalah 64; oleh karena itu, terdapat beberapa redundansi dalam kode genetik, dengan beberapa asam amino ditentukan oleh lebih dari satu kodon.[34] Gen yang disandi dalam DNA pertama-tama ditranskripsikan menjadi pra-RNA duta (mRNA) oleh protein seperti RNA polimerase. Kebanyakan organisme kemudian memproses pra-mRNA (juga dikenal sebagai transkrip primer) menggunakan berbagai bentuk modifikasi pascatranskripsi untuk membentuk mRNA yang matang, yang kemudian digunakan sebagai templat untuk sintesis protein oleh ribosom. Pada prokariota, mRNA dapat digunakan segera setelah diproduksi atau diikat oleh ribosom setelah menjauh dari nukleoid. Sebaliknya, eukariota membuat mRNA di inti sel dan kemudian mentranslokasikannya melewati membran inti ke dalam sitoplasma, tempat sintesis protein kemudian terjadi. Tingkat sintesis protein pada prokariota lebih tinggi daripada eukariota dan dapat mencapai hingga 20 asam amino per detik.[35]
Proses sintesis protein dari cetakan mRNA dikenal sebagai translasi. Selanjutnya, mRNA dimuat ke ribosom dan dibaca tiga nukleotida sekaligus dengan mencocokkan setiap kodon dengan antikodon pasangan basa yang terletak pada molekul RNA transfer (tRNA), yang membawa asam amino yang sesuai dengan kodon yang dikenalinya. Enzim sintetase tRNA-aminoasil "mengisi" molekul tRNA dengan asam amino yang benar. Polipeptida yang sedang terbentuk sering disebut rantai yang baru lahir. Protein selalu disintesis dari N-terminus ke C-terminus.[34]
Ukuran protein yang disintesis dapat diukur dengan jumlah asam amino yang dikandungnya dan dengan total massa molekulnya, yang biasanya dilaporkan dalam satuan dalton (identik dengan satuan massa atom), atau satuan turunan kilodalton (kDa). Ukuran rata-rata protein makin meningkat dari arkea, bakteri, dan eukariota (masing-masing 283, 311, 438 residu amino dan 31, 34, 49 kDa) karena lebih banyak domain protein yang menyusun protein dalam organisme yang lebih tinggi.[36] Misalnya protein khamir rata-rata memiliki panjang 466 asam amino dan massa 53 kDa. Protein terbesar yang diketahui adalah titin, komponen dari sarkomer otot, dengan massa molekul hampir 3.000 kDa dan panjang total hampir 27.000 asam amino.[37]
Sintesis kimia
Protein pendek juga dapat disintesis secara kimiawi dengan kelompok metode yang dikenal sebagai sintesis peptida, yang mengandalkan teknik sintesis organik seperti ligasi kimiawi untuk menghasilkan peptida dalam jumlah besar.[38] Sintesis kimia memungkinkan untuk memasukkan asam amino non-alami ke dalam rantai polipeptida, seperti pelekatan probe fluoresens ke rantai samping asam amino.[39] Metode ini berguna dalam laboratorium biokimia dan biologi sel, meskipun umumnya tidak untuk aplikasi komersial. Sintesis kimiawi tidak efisien untuk polipeptida yang lebih panjang dari sekitar 300 asam amino, dan protein yang disintesis mungkin tidak siap mengambil struktur tersier aslinya. Kebanyakan metode sintesis kimia berlanjut dari C-terminus ke N-terminus, berlawanan dengan reaksi biologis.[40]
Struktur
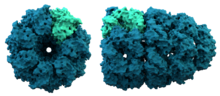

Sebagian besar protein terlipat menjadi struktur tiga dimensi yang unik. Bentuk alami suatu protein yang melipat dikenal dengan istilah konformasi asli.[41] Meskipun banyak protein dapat melipat tanpa bantuan dam hanya melalui sifat-sifat kimiawi asam amino mereka, sejumlah protein lain memerlukan bantuan protein pendamping untuk melipat menjadi kondisi aslinya.[42] Ahli biokimia sering merujuk pada empat aspek berbeda dari struktur protein.[43]
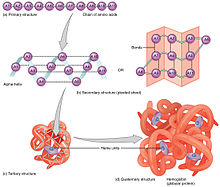
- Struktur primer, merupakan urutan asam amino yang dihubungkan melalui ikatan peptida (amida). Frederick Sanger merupakan ilmuwan yang berjasa dengan temuan metode penentuan deret asam amino pada protein, dengan penggunaan beberapa enzim protease yang mengiris ikatan antara asam amino tertentu menjadi fragmen peptida yang lebih pendek untuk dipisahkan lebih lanjut dengan bantuan kertas kromatografik. Urutan asam amino menentukan fungsi protein, pada tahun 1957, Vernon Ingram menemukan bahwa translokasi asam amino akan mengubah fungsi protein, dan lebih lanjut memicu mutasi genetik.
- Struktur sekunder, yaitu struktur tiga dimensi lokal dari berbagai rangkaian asam amino yang distabilkan oleh ikatan hidrogen. Contoh yang paling umum yaitu
- uliran-alfa (α-helix), berupa pilinan rantai asam amino-asam amino berbentuk seperti spiral;
- lempeng-beta (β-sheet), berupa lembaran-lembaran lebar yang tersusun dari sejumlah rantai asam amino yang saling terikat melalui ikatan hidrogen atau ikatan tiol (S–H);
- lekukan-beta (β-turn); dan
- lekukan-gama (γ-turn).[44]
- Struktur tersier, merupakan gabungan dari aneka ragam dari struktur sekunder dan menjadi bentuk keseluruhan satu molekul protein. Istilah "struktur tersier" sering digunakan sebagai sinonim dengan istilah lipatan. Struktur tersier inilah yang mengontrol fungsi dasar protein. Beberapa molekul protein dapat berinteraksi secara fisik tanpa ikatan kovalen membentuk oligomer yang stabil (misalnya dimer, trimer, atau kuartomer) dan membentuk struktur kuartener.
- Struktur kuartener, yaitu struktur yang dibentuk oleh beberapa molekul protein (rantai polipeptida). Dalam konteks ini, biasanya disebut subunit protein, yang berfungsi sebagai protein kompleks tunggal. Contoh yang terkenal adalah enzim Rubisco dan insulin.
- Struktur kuiner, yaitu karakteristik dari permukaan protein yang mengatur interior seluler yang padat. Struktur kuiner bergantung pada interaksi makromolekul yang bersifat sementara, tetapi penting, yang terjadi di dalam sel hidup.
Struktur primer protein bisa ditentukan dengan beberapa metode: (1) hidrolisis protein dengan asam kuat (misalnya, 6N HCl), lalu komposisi asam amino ditentukan dengan instrumen penganalisis asam amino, (2) analisis urutan dari ujung-N dilakukan dengan degradasi Edman, (3) kombinasi dari digesti dengan tripsin dan spektrometri massa, dan (4) penentuan massa molekuler dengan spektrometri massa.
Struktur sekunder bisa ditentukan dengan spektroskopi dikroisme sirkuler (CD) dan spektroskopi inframerah transformasi Fourier (FTIR).[45] Spektrum CD dari uliran-alfa menunjukkan dua absorbans negatif pada 208 dan 220 nm dan lempeng-beta menunjukkan satu puncak negatif sekitar 210–216 nm. Estimasi dari komposisi struktur sekunder dari protein bisa dikalkulasi dari spektrum CD. Pada spektrum FTIR, pita amida-I dari uliran-alfa berbeda dibandingkan dengan pita amida-I dari lempeng-beta. Jadi, komposisi struktur sekunder dari protein juga bisa diestimasi dari spektrum inframerah.
Protein bukanlah molekul yang sepenuhnya kaku. Selain tingkat struktur ini, protein dapat berubah di antara beberapa struktur terkait saat mereka menjalankan fungsinya. Dalam konteks penataan ulang fungsional ini, struktur tersier atau kuaterner biasanya disebut sebagai "konformasi", dan transisi di antara keduanya disebut perubahan konformasi. Perubahan tersebut sering kali disebabkan oleh pengikatan molekul substrat ke situs aktif enzim, atau wilayah fisik protein yang berpartisipasi dalam katalisis kimia. Dalam larutan, protein juga mengalami variasi struktur melalui getaran termal dan tumbukan dengan molekul lain.[46]

Secara informal, protein dapat dibagi menjadi tiga kelas utama yang berkorelasi dengan struktur tersier yang khas: protein globular, protein berserat, dan protein membran. Hampir semua protein globular dapat larut dan banyak di antaranya adalah enzim. Protein berserat sering kali bersifat struktural, seperti kolagen (komponen utama jaringan ikat) atau keratin (komponen protein rambut dan kuku). Protein membran sering berfungsi sebagai reseptor atau menyediakan saluran untuk molekul polar atau bermuatan untuk melewati membran sel.[47]
Dehidron merupakan kasus khusus dari ikatan hidrogen intramolekul di dalam protein, yang terlindung dengan buruk dari serangan air dan karenanya meningkatkan dehidrasinya sendiri.[48]
Domain protein
Struktur protein lainnya yang juga dikenal adalah domain, yaitu segmen protein yang melipat menjadi unit struktural yang berbeda. Struktur ini terdiri dari 40–350 asam amino. Protein sederhana umumnya hanya memiliki satu domain. Pada protein yang lebih kompleks, ada beberapa domain yang terlibat di dalamnya. Hubungan rantai polipeptida yang berperan di dalamnya akan menimbulkan sebuah fungsi baru berbeda dengan komponen penyusunnya. Bila struktur domain pada struktur kompleks ini berpisah, maka fungsi biologis masing-masing komponen domain penyusunnya tidak hilang. Inilah yang membedakan struktur domain dengan struktur kuartener. Pada struktur kuartener, setelah struktur kompleksnya berpisah, protein tersebut tidak fungsional. Domain biasanya memiliki fungsi spesifik, seperti aktivitas enzimatik (misalnya kinase) atau berfungsi sebagai modul pengikat (misalnya domain SH3 berikatan dengan urutan kaya prolin dalam protein lain).
Motif urutan
Urutan asam amino pendek dalam protein sering bertindak sebagai situs pengenalan untuk protein lain.[49] Misalnya, domain SH3 biasanya mengikat motif PxxP pendek (yaitu 2 prolin [P], yang dipisahkan oleh dua asam amino [x] yang tidak ditentukan, meskipun asam amino di sekitarnya dapat menentukan spesifisitas pengikatan yang tepat). Banyak motif semacam itu telah dikumpulkan dalam basis data Motif Linear Eukariotik (ELM).
Fungsi seluler
Protein adalah aktor utama di dalam sel, yang menjalankan tugas yang ditentukan oleh informasi yang disandi dalam gen.[50] Dengan pengecualian jenis RNA tertentu, sebagian besar molekul biologis lainnya adalah elemen yang relatif lembam dan dijadikan tempat protein bekerja. Protein menyusun setengah dari berat kering sel Escherichia coli, sedangkan makromolekul lain seperti DNA dan RNA masing-masing hanya berkontribusi sebesar 3% dan 20%.[51] Kumpulan protein yang diekspresikan dalam sel atau jenis sel tertentu dikenal sebagai proteoma.

Karakteristik utama protein yang juga memungkinkan beragam fungsi mereka adalah kemampuannya untuk mengikat molekul lain secara spesifik dan erat. Area protein yang bertanggung jawab untuk mengikat molekul lain dikenal sebagai situs pengikatan dan sering kali berupa cekungan atau "kantong" pada permukaan molekul. Kemampuan mengikat ini dimediasi oleh struktur tersier dari protein yang menentukan kantong situs pengikatan, dan oleh sifat kimiawi rantai samping asam amino di sekitarnya. Pengikatan protein bisa sangat ketat dan spesifik; sebagai contoh, protein penghambat ribonuklease berikatan dengan angiogenin manusia dengan konstanta disosiasi subfemtomolar (<10−15 M) tetapi tidak mengikat sama sekali dengan homolognya pada amfibi, yaitu onkonase (>1 M). Perubahan kimiawi yang sangat kecil seperti penambahan satu gugus metil ke pasangan-ikatan terkadang cukup untuk hampir menghilangkan pengikatan; misalnya enzim sintetase aminoasil-tRNA yang spesifik untuk asam amino valin, tidak mengikat rantai samping asam amino isoleusin yang sangat mirip.[52]
Protein dapat mengikat protein lain dan juga mengikat substrat molekul kecil. Ketika protein mengikat secara spesifik dengan salinan lain dari molekul yang sama, mereka dapat mengalami oligomerisasi untuk membentuk fibril; proses ini sering terjadi pada protein struktural yang terdiri dari monomer globular yang berikatan-sendiri untuk membentuk serat yang kaku. Interaksi protein-protein juga mengatur aktivitas enzimatik, mengendalikan perkembangan melalui siklus sel, dan memungkinkan perakitan kompleks protein besar yang melakukan banyak reaksi-terkait-serupa dengan fungsi biologis yang sama. Protein juga dapat mengikat atau bahkan diintegrasikan ke dalam membran sel. Kemampuan pasangan-ikatan untuk menginduksi perubahan konformasi protein memungkinkan pembangunan jaringan pensinyalan yang sangat kompleks.[53] Karena interaksi di antara protein bersifat reversibel dan sangat bergantung pada ketersediaan pasangan protein untuk membentuk agregat yang mampu melakukan rangkaian fungsi yang berbeda, studi tentang interaksi di antara protein tertentu adalah kunci untuk memahami aspek penting fungsi seluler, dan akhirnya sifat-sifat yang membedakan tipe sel tertentu.[54][55]
Enzim
Peran protein yang paling terkenal di dalam sel adalah sebagai enzim, yang mengkatalisasi reaksi kimia. Enzim biasanya sangat spesifik dan hanya mempercepat satu atau beberapa reaksi kimia. Enzim melakukan sebagian besar reaksi yang terlibat dalam metabolisme, serta memanipulasi DNA dalam berbagai proses seperti replikasi DNA, perbaikan DNA, dan transkripsi. Beberapa enzim bekerja pada protein lain untuk menambah atau menghilangkan gugus kimia dalam proses yang dikenal sebagai modifikasi pascatranslasi. Sekitar 4.000 reaksi dikatalisis oleh enzim.[56] Percepatan laju yang diberikan oleh katalisis enzimatis sering kali sangat besar, hingga peningkatan laju 1017 kali lipat dibandingkan reaksi tanpa katalisis dalam kasus orotat dekarboksilase (78 juta tahun tanpa enzim, 18 milidetik dengan enzim).[57]
Molekul yang terikat dan ditindaklanjuti oleh enzim disebut substrat. Meskipun enzim dapat terdiri dari ratusan asam amino, biasanya hanya sebagian kecil dari residu yang bersentuhan dengan substrat, dan fraksi yang lebih kecil lagi—rata-rata tiga hingga empat residu—yang terlibat langsung dalam katalisis. Area enzim yang mengikat substrat dan mengandung residu katalitik dikenal sebagai situs aktif.
Protein dirigen adalah anggota kelompok protein yang menentukan stereokimia senyawa yang disintesis oleh enzim lain.[58]
Pensinyalan sel dan pengikatan ligan

Banyak protein terlibat dalam proses pensinyalan sel dan transduksi sinyal. Beberapa protein, seperti insulin, merupakan protein ekstraseluler yang mengirimkan sinyal dari sel tempat mereka disintesis (yaitu sel pankreas) ke sel lain di jaringan yang jauh. Jenis lainnya adalah protein membran yang bertindak sebagai reseptor yang fungsi utamanya adalah mengikat molekul pemberi sinyal dan menginduksi respons biokimia di dalam sel. Banyak reseptor memiliki situs pengikatan yang terekspos pada permukaan sel dan domain efektor di dalam sel, yang mungkin memiliki aktivitas enzimatik atau mungkin mengalami perubahan konformasi yang dideteksi oleh protein lain di dalam sel.[59]
Antibodi adalah protein yang menjadi komponen dari sistem imun adaptif yang fungsi utamanya adalah mengikat antigen (zat asing di dalam tubuh) dan menargetkannya untuk dimusnahkan. Antibodi dapat disekresikan ke dalam lingkungan ekstraseluler atau berlabuh di membran sel B khusus yang dikenal sebagai sel plasma. Ketika enzim dibatasi dalam afinitas pengikatan terhadap substratnya oleh kebutuhannya untuk melakukan reaksi, antibodi tidak memiliki batasan seperti itu. Afinitas pengikatan antibodi ke targetnya sangat tinggi.[60]
Banyak protein transpor ligan mengikat biomolekul kecil tertentu dan membawanya ke lokasi lain di tubuh organisme multiseluler. Protein ini harus memiliki afinitas pengikatan yang tinggi jika ligannya terdapat dalam konsentrasi tinggi, tetapi juga harus melepaskan ligan saat berada pada konsentrasi rendah di jaringan target. Contoh protein pengikat ligan adalah hemoglobin, yang mengangkut oksigen dari paru-paru ke organ dan jaringan lain di semua vertebrata dan memiliki homolog serupa di setiap kerajaan biologis.[61] Lektin adalah protein pengikat gula yang sangat spesifik untuk bagian gula mereka. Lektin biasanya berperan dalam fenomena pengenalan biologis yang melibatkan sel dan protein.[62] Reseptor dan hormon adalah protein pengikat yang sangat spesifik.
Protein transmembran juga dapat berfungsi sebagai protein transpor ligan yang mengubah permeabilitas membran sel menjadi molekul dan ion kecil. Membran sendiri memiliki pusat yang hidrofobik sehingga molekul polar atau bermuatan tidak dapat berdifusi. Protein membran mengandung saluran internal yang memungkinkan molekul tersebut untuk masuk dan keluar sel. Banyak protein saluran ion dikhususkan agar hanya memilih ion tertentu; misalnya, saluran kalium dan natrium sering kali hanya memfasilitasi ion yang spesifik.[63]
Protein struktural
Protein struktural memberikan kekerasan dan kekakuan pada komponen biologis yang cair. Sebagian besar protein struktural merupakan protein berserat; misalnya kolagen dan elastin adalah komponen penting dari jaringan ikat seperti tulang rawan, sementara keratin ditemukan pada struktur keras atau berfilamen seperti rambut, kuku, bulu, tapak, dan beberapa cangkang hewan.[64] Beberapa protein globular juga dapat memiliki fungsi struktural, misalnya aktin dan tubulin yang bersifat globular dan dapat larut sebagai monomer, tetapi berpolimerisasi untuk membentuk serat kaku dan panjang yang membentuk sitoskeleton, yang memungkinkan sel untuk mempertahankan bentuk dan ukurannya.
Protein lain yang berfungsi struktural adalah protein motorik seperti miosin, kinesin, dan dinein, yang mampu menghasilkan gaya mekanis. Protein-protein ini sangat penting untuk motilitas seluler pada organisme bersel tunggal dan sperma pada banyak organisme multisel yang bereproduksi secara seksual. Mereka juga menghasilkan kekuatan yang digunakan dalam kontraksi otot,[65] serta memainkan peran penting dalam transportasi intraseluler.
Metode studi
Aktivitas dan struktur protein dapat diperiksa secara in vitro, in vivo, dan in silico. Studi in vitro tentang protein yang dimurnikan dalam lingkungan terkontrol berguna untuk mempelajari bagaimana protein menjalankan fungsinya. Misalnya, studi kinetika enzim mengeksplorasi mekanisme kimiawi dari aktivitas katalitik enzim dan afinitas relatifnya terhadap berbagai kemungkinan molekul substrat. Sebaliknya, percobaan in vivo dapat memberikan informasi tentang peran fisiologis protein dalam konteks sel atau bahkan organisme secara keseluruhan. Studi in silico menggunakan metode komputasi untuk mempelajari protein.
Pemurnian protein
Untuk melakukan analisis in vitro, protein harus dimurnikan dari komponen seluler lainnya. Proses ini biasanya dimulai dengan lisis sel, ketika membran sel terganggu dan isi internalnya dilepaskan ke dalam larutan yang dikenal sebagai lisat mentah. Campuran yang dihasilkan dapat dimurnikan menggunakan ultrasentrifugasi, yang memfraksinasi berbagai komponen seluler menjadi fraksi yang mengandung protein yang dapat larut; membran lipid dan protein; organel seluler, dan asam nukleat. Pengendapan dengan metode yang dikenal sebagai pengendapan terinduksi-garam dapat memusatkan protein dari lisat ini. Berbagai jenis kromatografi kemudian digunakan untuk mengisolasi protein yang diinginkan berdasarkan sifat-sifat seperti berat molekul, muatan bersih, dan afinitas pengikatan.[66] Tingkat pemurnian dapat dipantau dengan menggunakan berbagai jenis elektroforesis gel jika berat molekul dan titik isoelektrik protein yang diinginkan diketahui, dengan spektroskopi jika protein memiliki fitur spektroskopi yang dapat dibedakan, atau dengan uji enzim jika protein memiliki aktivitas enzimatik. Selain itu, protein dapat diisolasi sesuai dengan muatannya menggunakan pemfokusan listrik.[67]
Untuk protein alamiah, serangkaian langkah pemurnian mungkin diperlukan untuk mendapatkan protein yang cukup murni untuk aplikasi laboratorium. Untuk menyederhanakan proses ini, rekayasa genetika sering digunakan untuk menambahkan sifat kimiawi pada protein yang membuatnya lebih mudah untuk dimurnikan tanpa memengaruhi struktur atau aktivitasnya. Di sini, sebuah "penanda" yang terdiri dari urutan asam amino tertentu, biasanya serangkaian residu histidin (sebuah "penanda-His"), dilampirkan ke salah satu ujung protein. Akibatnya, ketika lisat dilewatkan ke kolom kromatografi yang mengandung nikel, residu histidin mengikat nikel dan menempel pada kolom, sementara komponen lisat yang tidak diberi tanda lewat tanpa hambatan. Sejumlah tag berbeda telah dikembangkan untuk membantu peneliti memurnikan protein tertentu dari campuran yang kompleks.[68]
Lokalisasi seluler
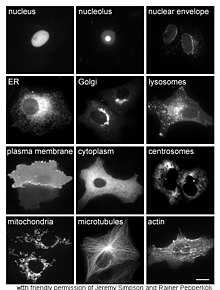
Studi tentang protein in vivo sering kali berkaitan dengan sintesis dan lokalisasi protein di dalam sel. Meskipun banyak protein intraseluler disintesis dalam sitoplasma dan protein-terikat-membran atau protein-tersekresi di retikulum endoplasma (RE), cara spesifik tentang bagaimana protein ditargetkan ke organel atau struktur seluler tertentu sering kali tidak jelas. Teknik yang berguna untuk menilai lokalisasi seluler adalah menggunakan rekayasa genetika untuk mengekspresikan di dalam sel, suatu protein fusi atau kimera yang terdiri dari protein alami yang diinginkan yang dihubungkan dengan "pelapor" seperti protein berpendar hijau (GFP).[69] Posisi protein yang menyatu di dalam sel dapat divisualisasikan dengan bersih dan efisien menggunakan mikroskop, seperti yang ditunjukkan pada gambar.[70]
Metode lain untuk menjelaskan lokasi seluler dari protein memerlukan penggunaan penanda kompartemen untuk daerah seperti RE, badan Golgi, lisosom atau vakuola, mitokondria, kloroplas, membran plasma, dan lainnya. Dengan penggunaan penanda berpendar (fluoresen) atau antibodi terhadap penanda, identifikasi lokalisasi protein yang diinginkan akan menjadi lebih mudah. Misalnya, imunofluoresensi tidak langsung akan memungkinkan kolokalisasi fluoresensi dan demonstrasi lokasi. Pewarna fluoresen digunakan untuk memberi label pada kompartemen seluler untuk tujuan yang sama.[71]
Kemungkinan lain juga ada. Misalnya imunohistokimia biasanya menggunakan antibodi terhadap satu protein yang diinginkan atau lebih, yang dikonjugasikan ke enzim yang menghasilkan sinyal bercahaya atau kromogenik yang dapat dibandingkan antarsampel, sehingga memungkinkan informasi lokalisasi. Teknik lain yang dapat diterapkan adalah kofraksionasi dalam gradien sukrosa (atau bahan lain) menggunakan sentrifugasi isopiknik.[72] Meskipun teknik ini tidak membuktikan kolokalisasi kompartemen dengan kepadatan yang diketahui dan protein yang diinginkan, teknik ini meningkatkan kemungkinan, dan lebih dapat diterima untuk penelitian skala besar.
Metode standar emas untuk lokalisasi seluler adalah mikroskop imunoelektron. Teknik ini juga menggunakan antibodi terhadap protein yang diinginkan, yang menggunakan teknik mikroskop elektron klasik. Sampel disiapkan untuk pemeriksaan mikroskopis elektron normal dan kemudian diberi antibodi terhadap protein yang diinginkan yang dikonjugasikan ke bahan yang sangat padat-elektro, biasanya emas. Hal ini memungkinkan untuk lokalisasi, baik detail ultrastruktur maupun protein yang diinginkan.[73]
Melalui penerapan rekayasa genetika lain yang dikenal sebagai mutagenesis-terarah-situs, para peneliti dapat mengubah urutan protein dan strukturnya, lokalisasi seluler, serta kerentanannya terhadap regulasi. Teknik ini bahkan memungkinkan penggabungan asam amino yang tidak alami ke dalam protein menggunakan tRNA yang dimodifikasi,[74] dan memungkinkan desain rasional protein baru dengan sifat baru.[75]
Proteomika
Jumlah komplemen protein yang ada pada suatu waktu dalam sel atau jenis sel dikenal sebagai proteoma, dan studi tentang kumpulan data berskala besar tersebut yaitu proteomika, yang dinamai sesuai dengan analoginya dalam genom yaitu genomika. Teknik eksperimental utama dalam proteomika meliputi elektroforesis 2D[76] yang memungkinkan pemisahan banyak protein, spektrometri massa[77] yang memungkinkan identifikasi protein dengan kecepatan tinggi dan pengurutan peptida dengan cepat (paling sering setelah pencernaan dalam gel), protein susunan-mikro yang memungkinkan deteksi jumlah berbagai protein yang ada dalam sel secara relatif, dan penapisan dua-hibrid yang memungkinkan eksplorasi sistematis interaksi protein-protein.[78] Jumlah komplemen yang mungkin secara biologis dari interaksi-interaksi semacam itu dikenal sebagai interaksioma. Upaya sistematis untuk menentukan struktur protein yang mewakili setiap kemungkinan lipatan dikenal sebagai genomika struktural.[79]
Bioinformatika
Berbagai macam metode komputasi telah dikembangkan untuk menganalisis struktur, fungsi, dan evolusi protein. Perkembangan metode-metode tersebut didorong oleh sejumlah besar data genomik dan proteomik yang tersedia untuk berbagai organisme, termasuk genom manusia. Tidak mungkin mempelajari semua protein secara eksperimental sehingga hanya sedikit protein yang menjadi sasaran eksperimen laboratorium. Sementara itu, alat komputasi digunakan untuk mengekstrapolasi ke protein yang serupa. Protein homolog dapat diidentifikasi secara efisien pada organisme yang berkerabat jauh melalui penjajaran urutan. Urutan-urutan genom dan gen dapat dicari dengan berbagai alat untuk properti tertentu. Alat pembuat profil urutan dapat menemukan situs enzim restriksi, rangka baca terbuka dalam urutan nukleotida, dan memprediksi struktur sekunder protein. Pohon filogenetika dapat dibuat dan hipotesis evolusi dikembangkan menggunakan perangkat lunak khusus seperti ClustalW untuk mengetahui nenek moyang organisme modern dan gen yang mereka ekspresikan. Bidang bioinformatika sangat diperlukan untuk analisis gen dan protein.
Penentuan struktur
Penemuan struktur tersier dari suatu protein, atau struktur kuaterner dari kompleks protein, dapat memberikan petunjuk penting tentang bagaimana protein tersebut menjalankan fungsinya dan bagaimana fungsi ini dapat dipengaruhi, misalnya dalam mendesain obat. Karena protein terlalu kecil untuk dilihat di bawah mikroskop cahaya, metode lain harus digunakan untuk menentukan strukturnya. Metode eksperimental yang umum meliputi kristalografi sinar-X dan spektroskopi NMR, keduanya dapat menghasilkan informasi struktural pada resolusi atomik. Eksperimen NMR mampu memberikan informasi dari mana subset jarak di antara pasangan atom dapat diperkirakan, dan kemungkinan konformasi akhir sebuah protein ditentukan dengan memecahkan masalah geometri jarak. Interferometri polarisasi ganda adalah metode analitik kuantitatif untuk mengukur konformasi protein secara keseluruhan dan perubahan konformasi akibat interaksi atau rangsangan lainnya. Dikroisme sirkuler adalah teknik laboratorium lain untuk menentukan komposisi untiran-alfa atau lembaran-beta internal dari protein. Mikroskop krioelektron digunakan untuk menghasilkan informasi struktural beresolusi rendah tentang kompleks protein yang sangat besar, termasuk virus yang telah dirakit;[80] varian yang dikenal sebagai kristalografi elektron juga dapat menghasilkan informasi resolusi tinggi dalam beberapa kasus, terutama untuk kristal protein membran dua dimensi.[81] Struktur yang diselesaikan biasanya disimpan di Bank Data Protein (PDB), sumber daya yang tersedia secara bebas mengenai data struktural dari ribuan protein yang dapat diperoleh dalam bentuk koordinat Cartesian untuk setiap atom dalam protein.[82]
Urutan gen lebih banyak diketahui dibandingkan struktur protein. Lebih jauh, himpunan struktur protein yang terselesaikan cenderung bias terhadap protein yang dapat dengan mudah mengalami kondisi yang diperlukan untuk kristalografi sinar-X, salah satu metode utama penentuan struktur protein. Secara khusus, protein globular secara komparatif mudah untuk mengkristal sebagai persiapan untuk kristalografi sinar-X. Sebaliknya, protein membran dan kompleks protein besar sulit untuk dikristalisasi dan kurang terwakili dalam PDB.[83] Genomika struktural telah berusaha untuk memperbaiki kekurangan ini dengan secara sistematis memecahkan struktur perwakilan dari kelas-kelas lipatan utama. Metode prediksi struktur protein mencoba mencari cara untuk menghasilkan struktur yang masuk akal untuk protein yang strukturnya belum ditentukan secara eksperimental.[84]
Prediksi dan simulasi struktur
Untuk melengkapi bidang genomika struktural, prediksi struktur protein mengembangkan model matematika protein yang efisien untuk memprediksi formasi molekul secara komputasi dalam teori, alih-alih mendeteksi struktur dengan observasi laboratorium.[85] Jenis prediksi struktur yang paling berhasil, yang dikenal sebagai pemodelan homologi, bergantung pada keberadaan struktur "templat" dengan kemiripan urutan terhadap protein yang dimodelkan; tujuan genomika struktural adalah memberikan representasi yang memadai dari struktur yang terselesaikan untuk memodelkan sebagian besar struktur yang tersisa.[86] Meskipun menghasilkan model yang akurat tetap menjadi tantangan ketika yang tersedia hanyalah struktur templat yang berkaitan jauh, disimpulkan bahwa penyelarasan urutan adalah penghambat dalam proses ini karena model yang cukup akurat dapat dihasilkan jika penyelarasan urutan yang "sempurna" diketahui.[87] Banyak metode prediksi struktur telah menyediakan informasi bagi bidang rekayasa protein, yang baru-baru ini muncul, ketika lipatan protein yang baru telah dirancang.[88] Masalah komputasi yang lebih kompleks yaitu prediksi interaksi antarmolekul, seperti dalam perkaitan molekuler dan prediksi interaksi protein-protein.[89]
Model matematika untuk mensimulasikan proses dinamis dari pelipatan dan pengikatan protein melibatkan mekanika molekuler, khususnya dinamika molekuler. Teknik Monte Carlo memfasilitasi komputasi, yang memanfaatkan kemajuan dalam komputasi paralel dan terdistribusi (misalnya proyek Folding@home[90] yang melakukan pemodelan molekuler pada GPU). Simulasi in silico menemukan lipatan domain protein uliran-alfa yang kecil seperti bagian-kepala protein vilin[91] dan protein aksesori HIV.[92] Metode hibrida yang menggabungkan dinamika molekul standar dengan matematika mekanika kuantum telah menjelajahi keadaan elektronik rhodopsin.[93]
Gangguan protein dan prediksi tidak terstruktur
Banyak protein (pada eukariota ~33%) mengandung segmen besar yang tidak terstruktur tetapi berfungsi secara biologis dan dapat diklasifikasikan sebagai protein yang tidak teratur secara intrinsik.[94] Oleh karena itu, memprediksi dan menganalisis kelainan protein merupakan bagian penting dari karakterisasi struktur protein.[95]
Analisis kimia
Jumlah kandungan nitrogen dari bahan organik terutama dibentuk oleh gugus amino dalam protein. Total Kjeldahl Nitrogen (TKN) adalah ukuran nitrogen yang banyak digunakan dalam analisis air (limbah), tanah, makanan, pakan, dan bahan organik secara umum. Seperti namanya, metode Kjeldahl diterapkan untuk menganalisisnya. Meskipun demikian, metode lain yang lebih sensitif juga tersedia.[96][97]
Nutrisi
Kebanyakan mikroorganisme dan tumbuhan dapat melakukan biosintesis untuk menghasilkan semua 20 asam amino standar, sedangkan hewan (termasuk manusia) harus memperoleh beberapa asam amino dari makanan.[51] Asam amino-asam amino yang tidak dapat disintesis sendiri oleh organisme disebut sebagai asam amino esensial. Enzim kunci yang menyintesis asam amino tertentu tidak terdapat pada hewan—seperti aspartokinase, yang mengkatalisis langkah pertama dalam sintesis lisin, metionin, dan treonin dari aspartat. Jika asam amino ada di lingkungan, mikroorganisme dapat menghemat energi dengan mengambil asam amino dari lingkungannya dan menurunkan jalur biosintetiknya.
Pada hewan, asam amino diperoleh melalui konsumsi makanan yang mengandung protein. Protein yang tertelan kemudian dipecah menjadi asam amino melalui pencernaan, yang biasanya melibatkan denaturasi protein melalui paparan asam dan hidrolisis oleh enzim yang disebut protease. Beberapa asam amino yang dicerna digunakan untuk biosintesis protein, sementara yang lain diubah menjadi glukosa melalui glukoneogenesis, atau dimasukkan ke dalam siklus asam sitrat. Penggunaan protein sebagai bahan bakar sangat penting dalam kondisi kelaparan karena memungkinkan protein tubuh digunakan untuk menyokong kehidupan, terutama protein yang ditemukan di otot.[98]
Pada hewan seperti anjing dan kucing, protein menjaga kesehatan dan kualitas kulit dengan mendorong pertumbuhan folikel rambut dan keratinisasi sehingga mengurangi kemungkinan munculnya bau busuk pada kulit.[99] Protein berkualitas buruk juga berperan dalam kesehatan saluran cerna dengan meningkatkan potensi perut kembung dan senyawa berbau pada anjing karena ketika protein mencapai usus besar dalam keadaan tidak tercerna, mereka difermentasi menghasilkan gas hidrogen sulfida, indol, dan skatol.[100] Anjing dan kucing mencerna protein hewani lebih baik dibandingkan protein nabati, tetapi produk hewani berkualitas rendah dicerna dengan buruk, termasuk kulit, bulu, dan jaringan ikat.[100]
Kekurangan protein bisa mengakibatkan kerontokan rambut (rambut terdiri dari 97-100% dari keratin) hingga busung lapar, penyakit kekurangan protein.[101] Kekurangan protein yang terus menerus menyebabkan marasmus dan berkibat kematian.
Studi dari Biokimiawan USA Thomas Osborne Lafayete Mendel, Profesor untuk biokimia di Yale, 1914, mengujicobakan protein konsumsi dari daging dan tumbuhan kepada kelinci. Satu grup kelinci-kelinci tersebut diberikan makanan protein hewani, sedangkan grup yang lain diberikan protein nabati. Dari eksperimennya didapati bahwa kelinci yang memperoleh protein hewani lebih cepat bertambah beratnya dari kelinci yang memperoleh protein nabati. Kemudian studi selanjutnya, oleh McCay dari Universitas Berkeley menunjukkan bahwa kelinci yang memperoleh protein nabati, lebih sehat dan hidup dua kali lebih lama.[butuh rujukan]
Referensi
- ^ Winarno, F. G. (1997). Kimia pangan dan gizi. semarang: PT Gramedia.
- ^ Thomas Burr Osborne (1909): The Vegetable Proteins , History pp 1 to 6, dari archive.org
- ^ Mulder GJ (1838). "Sur la composition de quelques substances animales". Bulletin des Sciences Physiques et Naturelles en Néerlande: 104.
- ^ Harold, Hartley (1951). "Origin of the Word 'Protein.'". Nature. 168 (4267): 244. Bibcode:1951Natur.168..244H. doi:10.1038/168244a0. PMID 14875059.
- ^ a b c Perrett D (August 2007). "From 'protein' to the beginnings of clinical proteomics". Proteomics: Clinical Applications. 1 (8): 720–38. doi:10.1002/prca.200700525. PMID 21136729.
- ^ New Oxford Dictionary of English
- ^ Reynolds JA, Tanford C (2003). Nature's Robots: A History of Proteins (Oxford Paperbacks). New York, New York: Oxford University Press. hlm. 15. ISBN 978-0-19-860694-9.
- ^ Reynolds and Tanford (2003).
- ^ Bischoff TL, Voit C (1860). Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt (dalam bahasa Jerman). Leipzig, Heidelberg.
- ^ "Hofmeister, Franz". encyclopedia.com. Diarsipkan dari versi asli tanggal 5 April 2017. Diakses tanggal 4 April 2017.
- ^ "Protein, section: Classification of protein". britannica.com. Diarsipkan dari versi asli tanggal 4 April 2017. Diakses tanggal 4 April 2017.
- ^ Sumner JB (1926). "The isolation and crystallization of the enzyme urease. Preliminary paper". Journal of Biological Chemistry. 69 (2): 435–41. Diarsipkan dari versi asli (PDF) tanggal 2011-03-25. Diakses tanggal 2011-01-16.
- ^ Pauling L, Corey RB (May 1951). "Atomic coordinates and structure factors for two helical configurations of polypeptide chains" (PDF). Proceedings of the National Academy of Sciences of the United States of America. 37 (5): 235–40. Bibcode:1951PNAS...37..235P. doi:10.1073/pnas.37.5.235. PMC 1063348
 . PMID 14834145. Diarsipkan dari versi asli (PDF) tanggal 2012-11-28. Diakses tanggal 2009-04-14.
. PMID 14834145. Diarsipkan dari versi asli (PDF) tanggal 2012-11-28. Diakses tanggal 2009-04-14.
- ^ Kauzmann W (May 1956). "Structural factors in protein denaturation". Journal of Cellular Physiology. 47 (Suppl 1): 113–31. doi:10.1002/jcp.1030470410. PMID 13332017.
- ^ Kauzmann W (1959). "Some factors in the interpretation of protein denaturation". Advances in Protein Chemistry Volume 14. Advances in Protein Chemistry. 14. hlm. 1–63. doi:10.1016/S0065-3233(08)60608-7. ISBN 978-0-12-034214-3. PMID 14404936.
- ^ Kalman SM, Linderstrøm-Lang K, Ottesen M, Richards FM (February 1955). "Degradation of ribonuclease by subtilisin". Biochimica et Biophysica Acta. 16 (2): 297–99. doi:10.1016/0006-3002(55)90224-9. PMID 14363272.
- ^ Sanger F (1949). "The terminal peptides of insulin". The Biochemical Journal. 45 (5): 563–74. doi:10.1042/bj0450563. PMC 1275055
 . PMID 15396627.
. PMID 15396627.
- ^ Sanger F. (1958), Nobel lecture: The chemistry of insulin (PDF), Nobelprize.org, diarsipkan dari versi asli (PDF) tanggal 2013-01-05, diakses tanggal 2016-02-09
- ^ Muirhead H, Perutz MF (August 1963). "Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution". Nature. 199 (4894): 633–38. Bibcode:1963Natur.199..633M. doi:10.1038/199633a0. PMID 14074546.
- ^ Kendrew JC, Bodo G, Dintzis HM, Parrish RG, Wyckoff H, Phillips DC (March 1958). "A three-dimensional model of the myoglobin molecule obtained by x-ray analysis". Nature. 181 (4610): 662–66. Bibcode:1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261.
- ^ "RCSB Protein Data Bank". Diarsipkan dari versi asli tanggal 2015-04-18. Diakses tanggal 2017-01-19.
- ^ Zhou ZH (April 2008). "Towards atomic resolution structural determination by single-particle cryo-electron microscopy". Current Opinion in Structural Biology. 18 (2): 218–28. doi:10.1016/j.sbi.2008.03.004. PMC 2714865
 . PMID 18403197.
. PMID 18403197.
- ^ Keskin O, Tuncbag N, Gursoy A (April 2008). "Characterization and prediction of protein interfaces to infer protein-protein interaction networks". Current Pharmaceutical Biotechnology. 9 (2): 67–76. doi:10.2174/138920108783955191. PMID 18393863.
- ^ Nelson DL, Cox MM (2005). Lehninger's Principles of Biochemistry (edisi ke-4). New York, New York: W. H. Freeman and Company.
- ^ Gutteridge A, Thornton JM (November 2005). "Understanding nature's catalytic toolkit". Trends in Biochemical Sciences. 30 (11): 622–29. doi:10.1016/j.tibs.2005.09.006. PMID 16214343.
- ^ Murray et al., p. 19.
- ^ Ardejani, Maziar S.; Powers, Evan T.; Kelly, Jeffery W. (2017). "Using Cooperatively Folded Peptides To Measure Interaction Energies and Conformational Propensities". Accounts of Chemical Research. 50 (8): 1875–82. doi:10.1021/acs.accounts.7b00195. ISSN 0001-4842. PMC 5584629
 . PMID 28723063.
. PMID 28723063.
- ^ Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland Pub. ISBN 978-0-8153-2305-1.
- ^ Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006). Harper's Illustrated Biochemistry. New York: Lange Medical Books/McGraw-Hill. ISBN 978-0-07-146197-9.
- ^ Van Holde KE, Mathews CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 978-0-8053-3931-4.
- ^ Milo R (December 2013). "What is the total number of protein molecules per cell volume? A call to rethink some published values". BioEssays. 35 (12): 1050–55. doi:10.1002/bies.201300066. PMC 3910158
 . PMID 24114984.
. PMID 24114984.
- ^ Beck M, Schmidt A, Malmstroem J, Claassen M, Ori A, Szymborska A, Herzog F, Rinner O, Ellenberg J, Aebersold R (November 2011). "The quantitative proteome of a human cell line". Molecular Systems Biology. 7: 549. doi:10.1038/msb.2011.82. PMC 3261713
 . PMID 22068332.
. PMID 22068332.
- ^ Wu L, Candille SI, Choi Y, Xie D, Jiang L, Li-Pook-Than J, Tang H, Snyder M (July 2013). "Variation and genetic control of protein abundance in humans". Nature. 499 (7456): 79–82. Bibcode:2013Natur.499...79W. doi:10.1038/nature12223. PMC 3789121
 . PMID 23676674.
. PMID 23676674.
- ^ a b van Holde and Mathews, pp. 1002–42.
- ^ Dobson CM (2000). "The nature and significance of protein folding". Dalam Pain RH. Mechanisms of Protein Folding. Oxford, Oxfordshire: Oxford University Press. hlm. 1–28. ISBN 978-0-19-963789-8.
- ^ Kozlowski LP (January 2017). "Proteome-pI: proteome isoelectric point database". Nucleic Acids Research. 45 (D1): D1112–D1116. doi:10.1093/nar/gkw978. PMC 5210655
 . PMID 27789699.
. PMID 27789699.
- ^ Fulton AB, Isaacs WB (April 1991). "Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis". BioEssays. 13 (4): 157–61. doi:10.1002/bies.950130403. PMID 1859393.
- ^ Bruckdorfer T, Marder O, Albericio F (February 2004). "From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future". Current Pharmaceutical Biotechnology. 5 (1): 29–43. doi:10.2174/1389201043489620. PMID 14965208.
- ^ Schwarzer D, Cole PA (December 2005). "Protein semisynthesis and expressed protein ligation: chasing a protein's tail". Current Opinion in Chemical Biology. 9 (6): 561–69. doi:10.1016/j.cbpa.2005.09.018. PMID 16226484.
- ^ Kent SB (February 2009). "Total chemical synthesis of proteins". Chemical Society Reviews. 38 (2): 338–51. doi:10.1039/b700141j. PMID 19169452.
- ^ Murray et al., p. 36.
- ^ Murray et al., p. 37.
- ^ Murray et al., pp. 30–34.
- ^ Paustian T. 2001. Protein Structure. University of Wisconsin-Madison. http://lecturer.ukdw.ac.id/dhira/BacterialStructure/Proteins.html Diarsipkan 2010-03-25 di Wayback Machine.. Diakses pada 5 Mei 2010.
- ^ Pribic R, Stokkum van IH, Chapman D, Haris PI, Bloemendal M. 1993. Protein secondary structure from Fourier transform infrared and/or circular dichroism spectra. Anal Biochem 214(2):366-78.
- ^ van Holde and Mathews, pp. 368–75.
- ^ van Holde and Mathews, pp. 165–85.
- ^ Fernández A, Scott R (September 2003). "Dehydron: a structurally encoded signal for protein interaction". Biophysical Journal. 85 (3): 1914–28. Bibcode:2003BpJ....85.1914F. doi:10.1016/S0006-3495(03)74619-0. PMC 1303363
 . PMID 12944304.
. PMID 12944304.
- ^ Davey NE, Van Roey K, Weatheritt RJ, Toedt G, Uyar B, Altenberg B, Budd A, Diella F, Dinkel H, Gibson TJ (January 2012). "Attributes of short linear motifs". Molecular BioSystems. 8 (1): 268–81. doi:10.1039/c1mb05231d. PMID 21909575.
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004). Molecular Cell Biology (edisi ke-5th). New York, New York: WH Freeman and Company.
- ^ a b Voet D, Voet JG. (2004). Biochemistry Vol 1 3rd ed. Wiley: Hoboken, NJ.
- ^ Sankaranarayanan R, Moras D (2001). "The fidelity of the translation of the genetic code". Acta Biochimica Polonica. 48 (2): 323–35. doi:10.18388/abp.2001_3918
 . PMID 11732604.
. PMID 11732604.
- ^ van Holde dan Mathews, pp. 830–49.
- ^ Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA (June 2009). "Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?". BioEssays. 31 (6): 629–41. doi:10.1002/bies.200800138. PMID 19382224.
- ^ Samarin S, Nusrat A (January 2009). "Regulation of epithelial apical junctional complex by Rho family GTPases". Frontiers in Bioscience. 14 (14): 1129–42. doi:10.2741/3298. PMID 19273120.
- ^ Bairoch A (January 2000). "The ENZYME database in 2000" (PDF). Nucleic Acids Research. 28 (1): 304–05. doi:10.1093/nar/28.1.304. PMC 102465
 . PMID 10592255. Diarsipkan dari versi asli (PDF) tanggal June 1, 2011.
. PMID 10592255. Diarsipkan dari versi asli (PDF) tanggal June 1, 2011.
- ^ Radzicka A, Wolfenden R (January 1995). "A proficient enzyme". Science. 267 (5194): 90–3. Bibcode:1995Sci...267...90R. doi:10.1126/science.7809611. PMID 7809611.
- ^ Pickel B, Schaller A (October 2013). "Dirigent proteins: molecular characteristics and potential biotechnological applications". Applied Microbiology and Biotechnology. 97 (19): 8427–38. doi:10.1007/s00253-013-5167-4. PMID 23989917.
- ^ Branden dan Tooze, pp. 251–281.
- ^ van Holde and Mathews, pp. 247–50.
- ^ van Holde and Mathews, pp. 220–29.
- ^ Rüdiger H, Siebert HC, Solís D, Jiménez-Barbero J, Romero A, von der Lieth CW, Diaz-Mariño T, Gabius HJ (April 2000). "Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets". Current Medicinal Chemistry. 7 (4): 389–416. doi:10.2174/0929867003375164. PMID 10702616.
- ^ Branden and Tooze, pp. 232–34.
- ^ van Holde and Mathews, pp. 178–81.
- ^ van Holde and Mathews, pp. 258–64, 272.
- ^ Murray et al., pp. 21–24.
- ^ Hey J, Posch A, Cohen A, Liu N, Harbers A (2008). "Fractionation of complex protein mixtures by liquid-phase isoelectric focusing". 2D PAGE: Sample Preparation and Fractionation. Methods in Molecular Biology. Methods in Molecular Biology™. 424. hlm. 225–39. doi:10.1007/978-1-60327-064-9_19. ISBN 978-1-58829-722-8. PMID 18369866.
- ^ Terpe K (January 2003). "Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems". Applied Microbiology and Biotechnology. 60 (5): 523–33. doi:10.1007/s00253-002-1158-6. PMID 12536251.
- ^ Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (August 2008). "Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes". Current Protein & Peptide Science. 9 (4): 338–69. doi:10.2174/138920308785132668. PMC 2904242
 . PMID 18691124.
. PMID 18691124.
- ^ Yuste R (December 2005). "Fluorescence microscopy today". Nature Methods. 2 (12): 902–4. doi:10.1038/nmeth1205-902. PMID 16299474.
- ^ Margolin W (January 2000). "Green fluorescent protein as a reporter for macromolecular localization in bacterial cells". Methods. 20 (1): 62–72. doi:10.1006/meth.1999.0906. PMID 10610805.
- ^ Walker JH, Wilson K (2000). Principles and Techniques of Practical Biochemistry. Cambridge, UK: Cambridge University Press. hlm. 287–89. ISBN 978-0-521-65873-7.
- ^ Mayhew TM, Lucocq JM (August 2008). "Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review". Histochemistry and Cell Biology. 130 (2): 299–313. doi:10.1007/s00418-008-0451-6. PMC 2491712
 . PMID 18553098.
. PMID 18553098.
- ^ Hohsaka T, Sisido M (December 2002). "Incorporation of non-natural amino acids into proteins". Current Opinion in Chemical Biology. 6 (6): 809–15. doi:10.1016/S1367-5931(02)00376-9. PMID 12470735.
- ^ Cedrone F, Ménez A, Quéméneur E (August 2000). "Tailoring new enzyme functions by rational redesign". Current Opinion in Structural Biology. 10 (4): 405–10. doi:10.1016/S0959-440X(00)00106-8. PMID 10981626.
- ^ Görg A, Weiss W, Dunn MJ (December 2004). "Current two-dimensional electrophoresis technology for proteomics". Proteomics. 4 (12): 3665–85. doi:10.1002/pmic.200401031. PMID 15543535.
- ^ Conrotto P, Souchelnytskyi S (September 2008). "Proteomic approaches in biological and medical sciences: principles and applications". Experimental Oncology. 30 (3): 171–80. PMID 18806738.
- ^ Koegl M, Uetz P (December 2007). "Improving yeast two-hybrid screening systems". Briefings in Functional Genomics & Proteomics. 6 (4): 302–12. doi:10.1093/bfgp/elm035
 . PMID 18218650. Diarsipkan dari versi asli tanggal 2017-09-11. Diakses tanggal 2017-07-23.
. PMID 18218650. Diarsipkan dari versi asli tanggal 2017-09-11. Diakses tanggal 2017-07-23.
- ^ Zhang C, Kim SH (February 2003). "Overview of structural genomics: from structure to function". Current Opinion in Chemical Biology. 7 (1): 28–32. doi:10.1016/S1367-5931(02)00015-7. PMID 12547423. Diarsipkan dari versi asli tanggal 2018-11-19. Diakses tanggal 2019-06-29.
- ^ Branden and Tooze, pp. 340–41.
- ^ Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T (December 2005). "Lipid-protein interactions in double-layered two-dimensional AQP0 crystals". Nature. 438 (7068): 633–38. Bibcode:2005Natur.438..633G. doi:10.1038/nature04321. PMC 1350984
 . PMID 16319884.
. PMID 16319884.
- ^ Standley DM, Kinjo AR, Kinoshita K, Nakamura H (July 2008). "Protein structure databases with new web services for structural biology and biomedical research". Briefings in Bioinformatics. 9 (4): 276–85. doi:10.1093/bib/bbn015
 . PMID 18430752. Diarsipkan dari versi asli tanggal 2013-04-15. Diakses tanggal 2009-04-13.
. PMID 18430752. Diarsipkan dari versi asli tanggal 2013-04-15. Diakses tanggal 2009-04-13.
- ^ Walian P, Cross TA, Jap BK (2004). "Structural genomics of membrane proteins". Genome Biology. 5 (4): 215. doi:10.1186/gb-2004-5-4-215. PMC 395774
 . PMID 15059248.
. PMID 15059248.
- ^ Sleator RD (2012). "Prediction of protein functions". Functional Genomics. Methods in Molecular Biology. 815. hlm. 15–24. doi:10.1007/978-1-61779-424-7_2. ISBN 978-1-61779-423-0. PMID 22130980.
- ^ Zhang Y (June 2008). "Progress and challenges in protein structure prediction". Current Opinion in Structural Biology. 18 (3): 342–48. doi:10.1016/j.sbi.2008.02.004. PMC 2680823
 . PMID 18436442.
. PMID 18436442.
- ^ Xiang Z (June 2006). "Advances in homology protein structure modeling". Current Protein & Peptide Science. 7 (3): 217–27. doi:10.2174/138920306777452312. PMC 1839925
 . PMID 16787261.
. PMID 16787261.
- ^ Zhang Y, Skolnick J (January 2005). "The protein structure prediction problem could be solved using the current PDB library". Proceedings of the National Academy of Sciences of the United States of America. 102 (4): 1029–34. Bibcode:2005PNAS..102.1029Z. doi:10.1073/pnas.0407152101. PMC 545829
 . PMID 15653774.
. PMID 15653774.
- ^ Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (November 2003). "Design of a novel globular protein fold with atomic-level accuracy". Science. 302 (5649): 1364–68. Bibcode:2003Sci...302.1364K. doi:10.1126/science.1089427. PMID 14631033.
- ^ Ritchie DW (February 2008). "Recent progress and future directions in protein-protein docking". Current Protein & Peptide Science. 9 (1): 1–15. CiteSeerX 10.1.1.211.4946
 . doi:10.2174/138920308783565741. PMID 18336319.
. doi:10.2174/138920308783565741. PMID 18336319.
- ^ Scheraga HA, Khalili M, Liwo A (2007). "Protein-folding dynamics: overview of molecular simulation techniques". Annual Review of Physical Chemistry. 58: 57–83. Bibcode:2007ARPC...58...57S. doi:10.1146/annurev.physchem.58.032806.104614. PMID 17034338.
- ^ Zagrovic B, Snow CD, Shirts MR, Pande VS (November 2002). "Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing". Journal of Molecular Biology. 323 (5): 927–37. CiteSeerX 10.1.1.142.8664
 . doi:10.1016/S0022-2836(02)00997-X. PMID 12417204.
. doi:10.1016/S0022-2836(02)00997-X. PMID 12417204.
- ^ Herges T, Wenzel W (January 2005). "In silico folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field". Physical Review Letters. 94 (1): 018101. arXiv:physics/0310146
 . Bibcode:2005PhRvL..94a8101H. doi:10.1103/PhysRevLett.94.018101. PMID 15698135.
. Bibcode:2005PhRvL..94a8101H. doi:10.1103/PhysRevLett.94.018101. PMID 15698135.
- ^ Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M (August 2006). "Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II". Journal of the American Chemical Society. 128 (33): 10808–18. doi:10.1021/ja062082i. PMID 16910676.
- ^ Ward JJ, Sodhi JS, McGuffin LJ, Buxton BF, Jones DT (March 2004). "Prediction and functional analysis of native disorder in proteins from the three kingdoms of life". Journal of Molecular Biology. 337 (3): 635–45. doi:10.1016/j.jmb.2004.02.002. PMID 15019783.
- ^ Tompa, Peter; Fersht, Alan (18 November 2009). Structure and Function of Intrinsically Disordered Proteins. CRC Press. ISBN 978-1-4200-7893-0. Diarsipkan dari versi asli tanggal 19 April 2017. Diakses tanggal 19 October 2016.
- ^ "Muñoz-Huerta et al. (2013) A Review of Methods for Sensing the Nitrogen Status in Plants: Advantages, Disadvantages and Recent Advances". Diarsipkan dari versi asli tanggal 2020-12-31. Diakses tanggal 2020-12-14.
- ^ "Martin et al. (2002) Determination of soil organic carbon and nitrogen at thefield level using near-infrared spectroscopy". Diarsipkan dari versi asli tanggal 2020-11-05. Diakses tanggal 2020-12-14.
- ^ Brosnan JT (June 2003). "Interorgan amino acid transport and its regulation". The Journal of Nutrition. 133 (6 Suppl 1): 2068S–72S. doi:10.1093/jn/133.6.2068S
 . PMID 12771367.
. PMID 12771367.
- ^ Watson TD (1998). "Diet and skin disease in dogs and cats". The Journal of Nutrition. 128 (12 Suppl): 2783S–89S. doi:10.1093/jn/128.12.2783S. PMID 9868266.
- ^ a b Case LP, Daristotle L, Hayek MG, Raasch MF (2010). Canine and Feline Nutrition-E-Book: A Resource for Companion Animal Professionals. Elsevier Health Sciences.
- ^ Prasanna HA, Desai BLM, Rao MN. 1971. Detection of early protein-calorie malnutrition (pre-kwashiorkor) in population groups. British J Nutr 26:71-74.
Buku teks
- Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland Pub. ISBN 978-0-8153-2305-1.
- Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006). Harper's Illustrated Biochemistry. New York: Lange Medical Books/McGraw-Hill. ISBN 978-0-07-146197-9.
- Van Holde KE, Mathews CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 978-0-8053-3931-4.
Pranala luar
Basis data dan proyek
- Basis data protein NCBI Entrez
- Basis data struktur protein NCBI
- Basis data referensi protein manusia
- Proteinpedia manusia
- Folding@Home (Universitas Stanford)Diarsipkan 2012-09-08 di Wayback Machine.
- Bank Data Protein di Eropa (lihat pula PDBeQuips[pranala nonaktif permanen], artikel singkat dan panduan tentang struktur PDB yang menarik)
- Research Collaboratory for Structural Bioinformatics (lihat pula Molecule of the Month Diarsipkan 2020-07-24 di Wayback Machine., menampilkan catatan singkat tentang protein terpilih dari PDB)
- Proteopedia – Life in 3D: model 3D yang dapat dirotasi dan diperbesar dengan anotasi wiki untuk setiap struktur molekuler protein yang diketahui.
- UniProt, sumber daya universal protein
Situs web pendidikan dan panduan
- "Pengantar tentang Protein" dari HOPES (Huntington's Disease Outreach Project for Education at Stanford)
- Protein: Biogenesi hingga Degradasi – Perpustakaan Virtual Biokimia dan Biologi Sel
- Protein di britannica.com


















