LIMSpec Wiki
목차
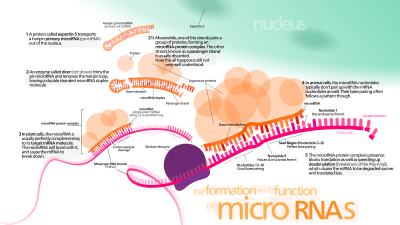

마이크로RNA(영어: microRNA, miRNA)는 식물, 동물, 바이러스 등에서 발견되는, 약 22개의 뉴클레오타이드로 구성된 작은 비발현 RNA 분자로, RNA 침묵과 전사 이후의 유전자 발현 조절 등의 기능을 한다.[1][2] 마이크로RNA는 식물과 동물 등 진핵 생물의 핵에 있는 DNA와, 게놈이 DNA에 기초한 특정 바이러스에 있는 바이러스성 DNA에 의해 암호화되면서, mRNA 분자에 있는 상호보완적인 염기쌍을 통해 기능을 한다. 그 결과, 이 mRNA 분자들은 하나 이상의 다음 과정들에 의해 침묵된다.[3]
마이크로RNA는 짧은 헤어핀을 형성하기 위해 스스로 꺾여 접히는 RNA 전사체의 영역으로부터 나온 마이크로RNA를 제외하고는 간섭 RNA(RNAi)경로의 작은 간섭 RNA(siRNA)와 유사하고, 반면 siRNA는 이중가닥 RNA의 더 긴 영역으로부터 나온다.[6] 인간의 게놈은 아마 많은 포유류의 세포에 풍부한 마이크로RNA를 약 1000개 이상 암호화하며,[7][8] 그것들은 인간과 다른 포유류의 유전자의 약 60%를 표적으로 할 것이다.[9][10]
마이크로RNA는 식물과 동물 모두에서 잘 보존되며, 필수적이고 진화적인 고대 유전자 조절 요소로 여겨진다.[11][12][13][14][15] 마이크로RNA 경로의 핵심 요소는 식물과 동물 사이에서 보존되지만, 각각의 마이크로RNA 레퍼토리는 다른 주요 활성 방식으로 독립적으로 나타난다.[16][17] 식물의 마이크로RNA는 주로 그들의 표적 mRNA와 거의 완벽한 쌍을 이루는데, 이것은 표적 전사체의 분리를 통해 유전자를 억제한다.[18] 반대로, 동물의 마이크로RNA는 5‘말단에 있는 6~8개 정도의 적은 뉴클레오타이드를 써서 그들의 표적을 인식할 수 있다.[19] 결합 조절은 동물의 마이크로RNA 조절의 특징이다.[20][21] 하나의 마이크로RNA는 수백 가지의 다른 표적 mRNA를 조절하고, 하나의 표적은 다수의 마이크로RNA에 의해 조절될 것이다.[22][23]
역사
마이크로RNA는 1993년에 Victor Ambros, Rosalind Lee, Rhonda Feinbaum가 예쁜 꼬마선충의 유충의 성장을 조절하는 lin-4 유전자를 연구하던 중에 처음 발견되었다.[24] 그들은 lin-4 유전자를 격리시켰을 때, 단백질을 암호화하는 mRNA를 생산하는 대신에 암호화되지 않은 RNA를 생산해내는 것을 발견하였고, 그것들 중 하나는 lin-14 mRNA의 3‘ UTR에 있는 많은 순서에 대해 부분적으로 상보적인 순서를 포함한 22개 뉴클레오타이드의 RNA였다.[25] 이 상보적 상태는 lin-14 mRNA가 LIN-14 단백질로 번역되는 것을 막게끔 했다. 당시에는 작은 lin-14 RNA가 선충의 특이한 성질로 여겨졌다. 2000년이 되어서야 두 번째 작은 RNA가 특징지어졌는데, 그것은 예쁜 꼬마선충의 나중에 있을 성장 상의 변화를 증진하기 위해 lin-41을 억제하는 let-7 RNA였다.[26] let-7 RNA는 곧 많은 종들에서 보존되는 것으로 발견되었고, let-7 RNA를 비롯한 더 많은 st RNA가 인간을 포함한 다양한 동물의 성장 시점을 조절할 것이라는 제안에 이르렀다.[27] 일 년 후에, lin-4와 let-7 RNA는 예쁜 꼬마선충, 초파리, 사람의 세포에 존재하는 작은 RNA들의 매우 큰 분류의 일부로 여겨졌다. 새롭게 발견되는 이러한 분류의 많은 RNA는 그들의 발현 패턴이 주로 성장 시점을 조절하는 역할에서 일관성이 없다는 것을 제외하고는 lin-4와 let-7과 유사했다. 이 시점에서 연구자들은 이러한 작은 조절 RNA의 분류를 지칭하기 위해 “마이크로 RNA”라는 용어를 처음 사용했다.[28][29][30]
RNA-induced silencing complex
miRNA는 RNA 발현의 억제에 관여한다. 이런 유전자 침묵은 miRNA가 mRNA를 퇴화시키거나 mRNA가 전사되는 것을 억제함으로써 이루어진다.
같이 보기
각주
- ↑ Ambros, V (Sep 16, 2004). "The functions of animal microRNAs". Nature 431 (7006): 350–5. doi:10.1038/nature02871. PMID 15372042.
- ↑ Bartel, DP (Jan 23, 2004). "MicroRNAs: genomics, biogenesis, mechanism, and function". Cell 116 (2): 281–97. doi:10.1016/S0092-8674(04)00045-5. PMID 14744438.
- ↑ Bartel DP (January 2009). "MicroRNAs: target recognition and regulatory functions". Cell 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ↑ Bartel DP (January 2009). "MicroRNAs: target recognition and regulatory functions". Cell 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ↑ Fabian, MR; Sonenberg, N; Filipowicz, W (2010). "Regulation of mRNA translation and stability by microRNAs". Annual review of biochemistry 79: 351–79. doi:10.1146/annurev-biochem-060308-103103. PMID 20533884.
- ↑ Bartel, DP (Jan 23, 2004). "MicroRNAs: genomics, biogenesis, mechanism, and function". Cell 116 (2): 281–97. doi:10.1016/S0092-8674(04)00045-5. PMID 14744438.
- ↑ Homo sapiens miRNAs in the miRBase at Manchester University
- ↑ Bentwich I, Avniel A, Karov Y, Aharonov R, Gilad S, Barad O, Barzilai A, Einat P, Einav U, Meiri E, Sharon E, Spector Y, Bentwich Z; Avniel; Karov; Aharonov; Gilad; Barad; Barzilai; Einat; Einav; Meiri; Sharon; Spector; Bentwich (July 2005). "Identification of hundreds of conserved and nonconserved human microRNAs". Nat. Genet. 37 (7): 766–70. doi:10.1038/ng1590. PMID 15965474.
- ↑ Lewis BP, Burge CB, Bartel DP (2005). "Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets". Cell 120 (1): 15–20. doi:10.1016/j.cell.2004.12.035. PMID 15652477.
- ↑ Friedman RC, Farh KK, Burge CB, Bartel DP; Farh; Burge; Bartel (January 2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ↑ Axtell, MJ; Bartel, DP (Jun 2005). "Antiquity of microRNAs and their targets in land plants". The Plant cell 17 (6): 1658–73. doi:10.1105/tpc.105.032185. PMC 1143068. PMID 15849273.
- ↑ Tanzer A, Stadler PF; Stadler (May 2004). "Molecular evolution of a microRNA cluster". J. Mol. Biol. 339 (2): 327–35. doi:10.1016/j.jmb.2004.03.065. PMID 15136036.
- ↑ Chen, Kevin; Rajewsky, Nikolaus (2007). "The evolution of gene regulation by transcription factors and microRNAs". Nature Reviews Genetics 8 (2): 93–103. doi:10.1038/nrg1990. PMID 17230196.
- ↑ Lee CT, Risom T, Strauss WM; Risom; Strauss (April 2007). "Evolutionary conservation of microRNA regulatory circuits: an examination of microRNA gene complexity and conserved microRNA-target interactions through metazoan phylogeny". DNA Cell Biol. 26 (4): 209–18. doi:10.1089/dna.2006.0545. PMID 17465887.
- ↑ Peterson, KJ; Dietrich, MR; McPeek, MA (Jul 2009). "MicroRNAs and metazoan macroevolution: insights into canalization, complexity, and the Cambrian explosion". BioEssays : news and reviews in molecular, cellular and developmental biology 31 (7): 736–47. doi:10.1002/bies.200900033. PMID 19472371.
- ↑ Shabalina SA, Koonin EV; Koonin (October 2008). "Origins and evolution of eukaryotic RNA interference". Trends in Ecology and Evolution. 10 (10): 578–587. doi:10.1016/j.tree.2008.06.005. PMC 2695246. PMID 18715673.
- ↑ Axtell, MJ; Westholm, JO; Lai, EC (2011). "Vive la différence: biogenesis and evolution of microRNAs in plants and animals". Genome Biology 12 (4): 221. doi:10.1186/gb-2011-12-4-221. PMC 3218855. PMID 21554756.
- ↑ Jones-Rhoades, MW; Bartel, DP; Bartel, B (2006). "MicroRNAS and their regulatory roles in plants". Annual review of plant biology 57: 19–53. doi:10.1146/annurev.arplant.57.032905.105218. PMID 16669754.
- ↑ Bartel DP (January 2009). "MicroRNAs: target recognition and regulatory functions". Cell 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ↑ Bartel DP (January 2009). "MicroRNAs: target recognition and regulatory functions". Cell 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ↑ Rajewsky, Nikolaus (2006). "microRNA target predictions in animals". Nature Genetics 38 (6s): S8–S13. doi:10.1038/ng1798.
- ↑ Friedman RC, Farh KK, Burge CB, Bartel DP; Farh; Burge; Bartel (January 2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ↑ Krek, Azra; Grün, Dominic; Poy, Matthew N; Wolf, Rachel; Rosenberg, Lauren; Epstein, Eric J; MacMenamin, Philip; da Piedade, Isabelle; Gunsalus, Kristin C; Stoffel, Markus; Rajewsky, Nikolaus (May 2005). "Combinatorial microRNA target predictions". Nature Genetics 37 (5): 495–500. doi:10.1038/ng1536. PMID 15806104.
- ↑ Lee RC, Feinbaum RL, Ambros V; Feinbaum; Ambros (December 1993). "The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14". Cell 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ↑ Lee RC, Feinbaum RL, Ambros V; Feinbaum; Ambros (December 1993). "The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14". Cell 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ↑ Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G; Slack; Basson; Pasquinelli; Bettinger; Rougvie; Horvitz; Ruvkun (February 2000). "The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans". Nature 403 (6772): 901–6. Bibcode:2000Natur.403..901R. doi:10.1038/35002607. PMID 10706289.
- ↑ Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, Hayward DC, Ball EE, Degnan B, Müller P, Spring J, Srinivasan A, Fishman M, Finnerty J, Corbo J, Levine M, Leahy P, Davidson E, Ruvkun G; Reinhart; Slack; Martindale; Kuroda; Maller; Hayward; Ball; Degnan; Müller; Spring; Srinivasan; Fishman; Finnerty; Corbo; Levine; Leahy; Davidson; Ruvkun (November 2000). "Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA". Nature 408 (6808): 86–9. doi:10.1038/35040556. PMID 11081512.
- ↑ Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T; Rauhut; Lendeckel; Tuschl (October 2001). "Identification of novel genes coding for small expressed RNAs". Science 294 (5543): 853–8. Bibcode:2001Sci...294..853L. doi:10.1126/science.1064921. PMID 11679670.
- ↑ Lau NC, Lim LP, Weinstein EG, Bartel DP; Lim; Weinstein; Bartel (October 2001). "An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans". Science 294 (5543): 858–62. Bibcode:2001Sci...294..858L. doi:10.1126/science.1065062. PMID 11679671.
- ↑ Lee RC, Ambros V; Ambros (October 2001). "An extensive class of small RNAs in Caenorhabditis elegans". Science 294 (5543): 862–4. Bibcode:2001Sci...294..862L. doi:10.1126/science.1065329. PMID 11679672.

















