Clinfowiki
Contidos
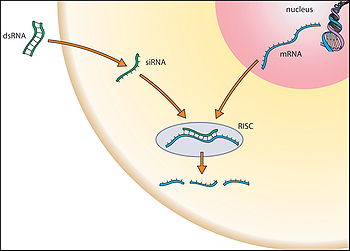
O ARN interferente pequeno, ARNip ou siRNA (small interfering RNA), tamén chamado ARN interferente curto ou ARN silenciador, é un tipo de ARN bicatenario de 20-25 nucleótidos de lonxitude, que ten diversas funcións biolóxicas, a principal das cales é a súa participación na interferencia por ARN (RNAi), na que estes ARN interfiren a expresión dun xene específico. Ademais desta función na vía da interferencia por ARN, o siRNA actúa tamén noutras vías relacionadas con dita interferencia; por exemplo, como mecanismo antiviral ou en darlle forma a estrutura da cromatina do xenoma; a complexidade destes procesos aínda non foi de todo aclarada.
Os siRNA foron descubertos polo grupo de David Baulcombe no Sainsbury Laboratory en Norwich, Inglaterra, como parte do silenciamento xénico postranscricional (PTGS) en plantas[1]. Pouco despois, en 2001, viuse que os siRNA sintéticos podían inducir interferencia (RNAi) en células de mamíferos grazas aos traballos do grupo de Thomas Tuschl[2]. Este descubrimento fixo crecer o interese en procurar o modo de aproveitar a RNAi nas investigacións biomédicas e no desenvolvemento de fármacos.
Estrutura
Os siRNA teñen unha estrutura ben definida: son ARN de dobre cadea (dsRNA) curtos de xeralmente de 21 nucleótidos (nt) con 2 nt extra non emparellados en cada extremo 3', que sobresaen.

Cada cadea ten un grupo fosfato 5' e un grupo hidroxilo (-OH) 3'. Esta estrutura é o resultado do procesamento feito por Dicer, un encima que converte os ARN bicatenarios longos ou os ARN forquita pequenos (shRNA) en siRNA.[3] Os siRNA poden tamén ser introducidos exoxenamente (artificialmente) nas células por varios métodos de transfección para producir o knockdown específico do xene que interese. Basicamente, calquera xene de secuencia coñecida, pode ser o branco destas técnicas, usando un siRNA de secuencia complementaria adecuadamente preparado. Isto fai dos siRNA unha importante ferramenta no estudo da función xénica e nos estudos de validación de certos fármacos na era posxenómica.
Indución da interferencia por ARN utilizando siRNAs ou os seus precursores

A transfección dun siRNA exóxeno pode causar problemas porque o efecto do knockdown do xene é só transitorio, especialmente en células en rápida división. Un xeito de superar este inconveniente é modificar o siRNA dun modo que lle permita ser expresado por un vector apropiado, como un plásmido. Isto faise por medio da introdución dun bucle entre as dúas cadeas, que produza un único transcrito, que poida ser procesado dando un siRNA funcional. Estes casetes de transcrición úsanse normalmente cun promotor de ARN polimerase III (por exemplo, U6 ou H1), que xeralmente dirixe a transcrición dos ARN nucleares pequenos (snRNA) (U6 está implicado no splicing; H1 é o compoñente ribonuclease da RNase P humana). Pénsase (aínda que a seguridade non é total) que o transcrito de siRNA resultante é despois procesado por Dicer.
Activación do ARN
Descubriuse recentemente que o ARN bicatenario (dsRNA) pode tamén activar a expresión xénica por un mecanismo chamado "activación xénica inducida por ARN pequenos" ou RNAa. Observouse que os promotores dos xenes diana dos dsRNA inducen unha potente activación transcricional dos xenes asociados a eles. A RNAa foi demostrada tamén en células humanas utilizando dsRNA sintéticos, denominados "ARN pequenos activadores" (saRNA). Actualmente non se sabe se a RNAa está conservada noutros organismos.[4]
Como evitar os efectos non específicos
Dado que a interferencia por ARN (RNAi) se cruza con outras vías biolóxicas, non debe sorprender que en ocasións a introdución experimental dun siRNA desencadee efectos non específicos. Cando unha célula de mamífero se atopa cun ARN de dobre cadea como son os siRNA, pode confundilo cun produto de procedencia viral e iniciar unha resposta inmune. Ademais, como os microARN (estruturalmente moi relacionados) modulan a expresión xénica en gran medida por interaccións de complementariedade de bases co seu ARNm obxectivo, a introdución dun siRNA pode causar un efecto non pretendido fóra do obxectivo procurado.
Inmunidade innata
A introdución de demasiado siRNA pode causar efectos non específicos debido á activación de respostas inmunes innatas. A maioría das evidencias coñecidas ata agora suxiren que isto é debido probablemente á activación do sensor PKR do ARN de dobre cadea, aínda que o xene I (RIG-I) inducible polo ácido retinoico pode estar implicado tamén[Cómpre referencia]. Tamén se descubriu a indución de citoquinas polo receptor 7 (TLR7) de tipo Toll. Un método moi prometedor para reducir os efectos non específicos é converter o siRNA en microARN. Os microARN están presentes naturalmente, e aproveitando esta vía endóxena debería ser posible conseguir un knockdown similar de xenes a concentracións comparativamente baixas dos siRNA resultantes. Isto debería minimizar os efectos non específicos.
Efectos "fóra do obxectivo"
Evitar os efectos "fóra do obxectivo" é outro reto para o uso dos siRNAs como ferramenta para o knockdown de xenes. A expresión dos xenes con complementariedade incompleta é inadvertidamente regulada á baixa polos siRNA (o siRNA actuaría como un microARN), o que coneva problemas na interpretación dos datos e na toxicidade potencial. Isto, porén, pode ser parcialmente contrarrestado deseñando experimentos de control apropiados, e estanse a desenvolver actualmente os algoritmos de deseño de siRNA para producir siRNAs libres de efectos fóra do obxectivo. A análise da expresión de todo o xenoma, por exemplo, por medio da tecnoloxía de micromatrices ou microarrays, pode utilizarse despois para verificar isto e para un ulterior refinamento dos algoritmos. Unha publicación de 2006 do laboratorio do Dr. Khvorova implica tramos de 6 ou 7 pares de base de lonxitude desde a posición 2 en adiante na unión complementaria do siRNA coas rexións 3'-UTR en xenes fóra do obxectivo.[5]
Posibles aplicacións terapéuticas
Dado que é posible bloquear calquera xene que interese por medio da interferencia (RNAi) realizada con siRNA, estas técnicas xeraron unha grande interese tanto en bioloxía básica[6] coma aplicada. Hai un crecente número de probas de RNAi de grande escala deseñadas para identificar os xenes importantes de diversos procesos biolóxicos. Como os procesos patolóxicos das enfermidades tamén dependen da actividade de múltiples xenes, espérase que nalgunhas situacións podería conseguirse un beneficio terapéutico bloqueando a actividade dun xene con siRNA.
Non obstante, aplicar interferencia (RNAi) por medio de siRNAs a animais vivos, especialmente a seres humanos, ten diversos perigos potenciais. Nos experimentos, os siRNA mostran unha efectividade diferente en distintos tipos de células dun modo que aínda non se coñece ben: algunhas células responden ben aos siRNA e mostran un potente knockdown xenético mentres que outras non mostran este efecto (mesmo a pesar de que a transfección fose eficiente).
Os resultados da fase I dos primeiros ensaios de RNAi terapéutica (indicada para a dexeneración muscular relacionada coa idade, tamén coñecida como AMD) sinalaban a finais de 2005 que os siRNA eran ben tolerados e tiñan unhas propiedades farmacocinéticas axeitadas.[7]
Outros ensaios indicaron que os siRNA dirixidos contra o virus Ébola poden ser efectivos como unha profilaxe postexposición en humanos, cunha supervivencia de ata o 100% en primates non humanos ante unha dose letal do virus Zaire Ebola, a estirpe máis letal dese virus.[8]
Notas
- ↑ Hamilton A, Baulcombe D (1999). "A species of small antisense RNA in posttranscriptional gene silencing in plants". Science 286 (5441): 950–2. PMID 10542148. doi:10.1126/science.286.5441.950.
- ↑ Elbashir S, Harborth J, Lendeckel W, Yalcin A, Weber K, Tuschl T (2001). "Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells". Nature 411 (6836): 494–988. PMID 11373684. doi:10.1038/35078107.
- ↑ Bernstein E, Caudy A, Hammond S, Hannon G (2001). "Role for a bidentate ribonuclease in the initiation step of RNA interference". Nature 409 (6818): 363–366. PMID 11201747. doi:10.1038/35053110.
- ↑ Li LC (2008). "Small RNA-Mediated Gene Activation". RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Caister Academic Press. ISBN 978-1-904455-25-7.
- ↑ Birmingham A, Anderson E, Reynolds A, Ilsley-Tyree D, Leake D, Fedorov Y, Baskerville S, Maksimova E, Robinson K, Karpilow J, Marshall W, Khvorova A (2006). "3' UTR seed matches, but not overall identity, are associated with RNAi off-targets". Nat Methods 3 (3): 199–204. PMID 16489337. doi:10.1038/nmeth854.
- ↑ Alekseev OM, Richardson RT, Alekseev O, O'Rand MG (2009). "Analysis of gene expression profiles in HeLa cells in response to overexpression or siRNA-mediated depletion of NASP". Reproductive Biology and Endocrinology 7: 45. PMC 2686705. PMID 19439102. doi:10.1186/1477-7827-7-45.
- ↑ Tansey B (11 August 2006). "Macular degeneration treatment interferes with RNA messages". San Francisco Chronicle.
- ↑ Postexposure protection of non-human primates against a lethal Ebola virus challenge with RNA interference: a proof-of-concept study Prof Thomas W Geisbert PhD,Amy CH Lee MSc,Marjorie Robbins PhD,Joan B Geisbert,Anna N Honko PhD,Vandana Sood MSc,Joshua C Johnson BSc,Susan de Jong PhD,Iran Tavakoli BSc,Adam Judge PhD,Lisa E Hensley PhD,Ian MacLachlan PhD The Lancet - 29 May 2010 ( Vol. 375, Issue 9729, Pages 1896-1905 ) DOI: 10.1016/S0140-6736(10)60357-1
Véxase tamén
Outros artigos
Ligazóns externas
- siRNAdb: base de datos de secuencias de siRNA
- DesiRM: Deseño de siRNA complementario e non complementario para o silenciamento de xenes.

















